PUBLICATION
육종학자와 병리학자들에게 식물의 면역, 특히 면역 신호 전달시스템을 활성화시키는 수용체(MAMP: microbe-associated molecular patterns)를 인식하는 세포 표면 단백질 또는 NLR(nucleotide–binding leucine-rich repeat receptors) 등 pathogen effector에 반응하는 세포내 단백질 등에 대해 지속적인 관심을 보여왔습니다.
개화식물의 유전체에서 발견된 수백개의 NLR유전자들은 병원체 인식 후 세포 사멸을 제어하는 inflammasome 유사체 또는 resistosome을 형성한다고 생각되어 새로운 병원체 저항성을 만들어 낼 수 있는 후보군으로써 연구되고 있습니다. 연구자들은 다양한 분류 수준에서 NLR 유전자의 inventory를 만드려고 시도하고 있으나, 유전자 패밀리의 다형성, 대립 유전자 및 구조적 변이의 패턴 및 광범위한 카피 수 변이를 갖는 cluster 등으로 어려움을 겪고 있습니다.
아래에 소개드릴 두 연구 팀은 resistance gene enrichment sequencing(RenSeq)과 SMRT 시퀀싱을 결합하여 이러한 과제를 성공적으로 극복했습니다.
A Species-Wide Inventory of NLR Genes and Alleles in Arabidopsis thaliana
Cell Volume 178, Issue 5, 22 August 2019, Pages 1260-1272.e14
https://doi.org/10.1016/j.cell.2019.07.038
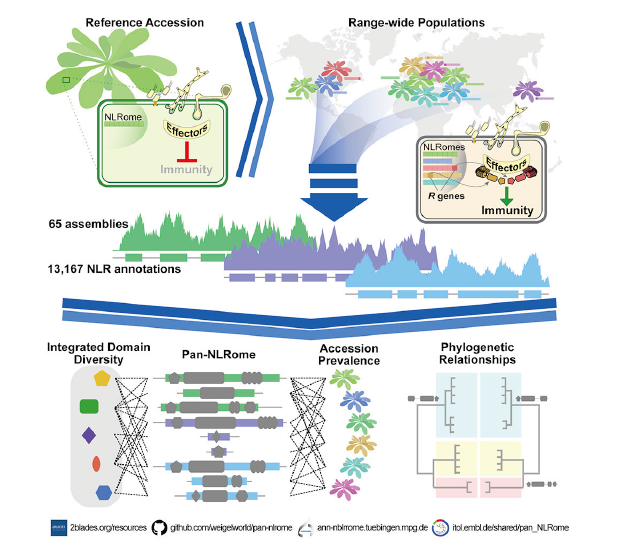
독일의 연합연구팀은 Arabidopsis thaliana 종의 유전자 전체에서의 NLR 서열을 거의 완성된 상태로 발표하였습니다.
핵심 NLR의 보완할 곳을 찾았을 뿐만아니라 통합된 도메인의 다양성, 새로운 도메인의 구조, non-core NLR 내의 polymorphism의 존재 여부 등을 밝혀내고, A. thaliana Col-0 참조 게놈에 매핑할 수 있었습니다.
Evolution of NLR resistance genes with non-canonical N-terminal domains in wild tomato species
bioRxiv preprint
https://doi.org/10.1101/786194
UC 버클리 팀은 토마토 (Solanum lycopersicum)의 NLRome에 대한 결과를 BioRxiv에 발표하였습니다.
다양한 병원균에 의한 200가지 이상의 질병에 시달리고 있는 이 중요 작물은 특히 재배하는 과정에서 토마토의 저항성(R) 유전자가 변화하고 유전적 다양성이 낮아 유전자 개선이 시급합니다. 과학자들은 다양한 서식지에서 병원균과 함께 진화해온 야생 토마토 종의 유전적 질병 저항력과 특히 R 유전자가 복합 병원균에 대한 내성이 더 강하다는 것을 밝혀내자고 하였으나 반복구간과 NLR의 물리적 구조를 밝혀내기에는 방법적 제한이 있었습니다.
본 연구팀은 PacBio SMRT sequencing(SMRT RenSeq)을 활용하여 18개 종에서 NLR를 밝혀냈습니다. 채워지지 않은 gap이 있거나 mis-assembly 되어 미완성으로 있었던 13개의 기존 annotation을 포함하여 128개 NLR의 annotation을 향상시켰습니다. RenSeq을 활용한 이러한 성과에 대하여 연구진은 다음과 같이 평가했습니다.
“We demonstrated that SMRT RenSeq is a cost-effective, efficient alternative to the whole genome sequencing. We also verified that SMRT RenSeq was capable of…resolving the complexity of NLRs and their clusters.”











