
감염된 숙주 내에서 바이러스의 복제 및 경쟁이 진행되면서 복잡한 변종 혼합물이 생성됩니다. 한 집단 내의 모든 변이를 분석하는 것은 진화, 유사종 역학, 약물 내성 및 면역 회피 등을 이해하는 데 중요합니다.
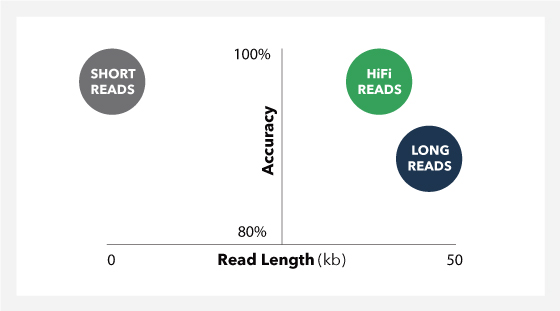
HiFi 시퀀싱은 최대 20kb 길이의 amplicon에서 99 % 이상의 정확한 read를 제공하여 phase variant를 직접 감지합니다.
- 복잡한 혼합물을 유사종 및 고유한 haplotype으로 분리 - 커뮤니티 내 또는 지리적인 영역 전체에서 숙주 내 바이러스 집단의 진화 및 계통을 추적 - 면역 회피 또는 약물 내성과 관련된 minor variant 식별 및 정량 - 대규모 바이러스 genome의 de novo assembly 완성
|
Spotlight: SMRT Sequencing of full-length HIV envelope
reveals complete compartmentalization of variants in brain versus all other
tissues


• SMRT sequencing 기술은 한 번에 수천 개의 긴 read를 생성 할 수 있기 때문에
뇌와 림프 조직에서 HIV 분리샘플 간의 관계를 연구 할 수 있는 좋은 기회를 |

Explore the microbial world with confidence
매우 정확하고 길이가 긴 HiFi read는 single-molecule 수준으로 분석이 가능
하므로,
full-length 16S rRNA sequencing , shotgun metagenome profiling 및 metagenome profiling에 적합합니다. - 경제적인 가격으로 full-length 16S rRNA
sequencing을 수행, 종 또는 균주 수준에서
community 구성을 분석 - 절감된 비용으로 효율적인 metagenome profiling을 수행, HiFi read 에서 6-8 개의
full-length gene 식별 - 6Gb의
HiFi 데이터로
multiplexed human fecal sample에서 최대 20 개의 고품질 metagenome –assembled genome (MAG) 생성 - 후성 유전체 데이터를 활용하여 밀접하게 관련된 균주의 contig 와 plasmid 를 연관분석 |

Callahan, B. J., et al. (2019). Nucleic Acids Research.
미생물/metagenome workflow의 성능을 평가하기 위해 다양한 크기와 세포벽 구성을 가진 gram negative 및 gram positive 박테리아와 효모로 구성된 모의 Communities 인 Zymo mock community를 사용하여 DNA를 추출하고 full-length 16S rRNA gene 영역을 증폭, PacBio SMRT library를 제작하여 Sequencing을 진행하였습니다. |
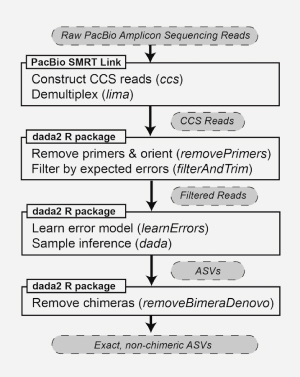
▲ Flowchart of computational methodology |

▲ Abundances of full-length 16S rRNA gene amplicon sequence variants (ASVs) detected in the Zymo mock community

Beaulaurier, J., et al. (2017). Nature
DOI: 10.1038/nbt.4037
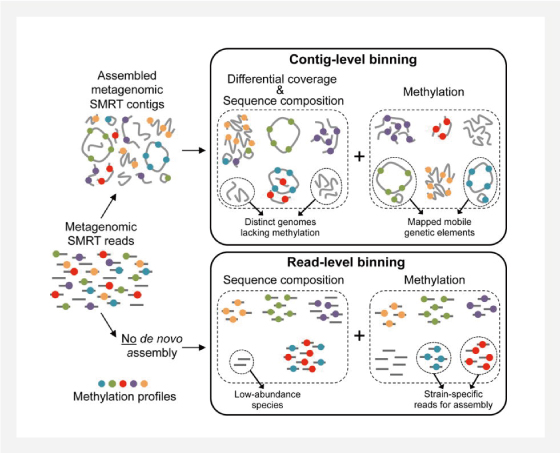
▲ Overview of metagenomic binning
using DNA methylation detected in SMRT long reads
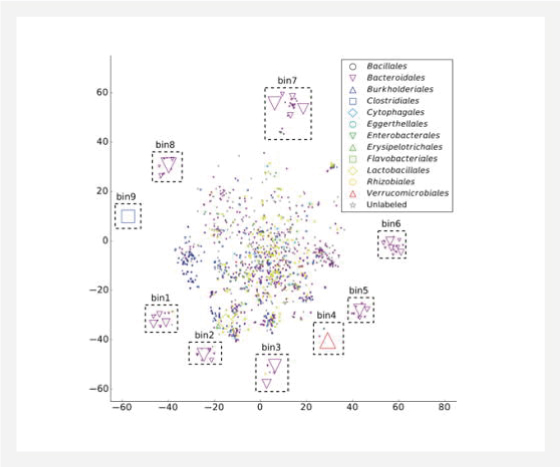
▲ Binning mouse gut microbiome
sequencing data

네가지 환경 sample에서 다양한 prokaryotes로부터 full?length에 가까운 rRNA operon amplicons을 대상으로 sequencing 하였습니다.
• Archaeal SILVA 데이터베이스와 비교하여 많은 novel한 것으로 추정되는 archaeal 23S rRNA gene sequences 을 생성했습니다.
• 이러한 long amplicon은 긴 (~ 1000 bp) 16S rRNA 유전자 단편을 통해 분류 학적 분류 중에 더 높은 resolution을 보이며 short amplicon 에 비해 near full?length 16S 와 23S rRNA gene sequences 을 결합한 정보가 훨씬 더 확실한 phylogeny 정보를 제공함을 보여주고 있습니다.
|
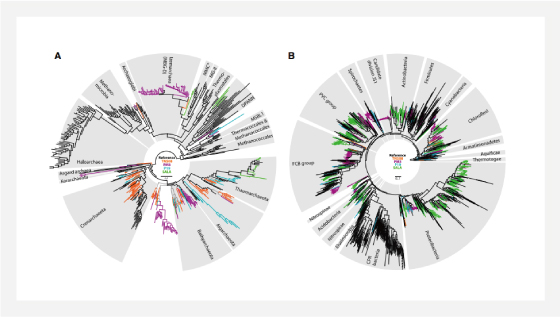
▲ Captured archaeal (A) and bacterial (B) phylogenetic diversity in the environmental samples
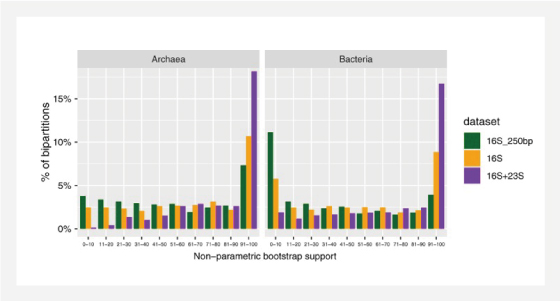
▲ Separate comparisons for Archaea
and bacteria data sets